Welcome!
This community is for professionals and enthusiasts of our products and services. Share and discuss the best content and new marketing ideas, build your professional profile and become a better marketer together.
基于機器學習翻譯,僅供參考。
練習2:查找CRISPR位點并檢查脫靶匹配度
除了感興趣的基因之外,gRNA序列可以結合基因組中的其他區域。Cas9也會影響這些基因組區域的序列。根據這些其他結合位點的位置,這可能會產生嚴重的意外后果。當選擇CRISPR位點時,我們通常感興趣的是基因組中gRNA序列可能與多少個其他位置結合以及這可能具有什么影響。Geneious中的Find CRISPR位點工具可以搜索非目標綁定,并使用此信息為每個CRISPR位點計算得分。
在第一個練習中,您使用Find CRISPR位點功能查找LYP1 CDS中的所有“GN(20)GG”位點。在本練習中,您還將檢查異常與釀酒酵母基因組的結合情況。
要做到這一點,首先需要創建一個您希望針對脫靶結合位點進行測試的序列數據庫 - 這通常是您感興趣生物組織的整個基因組,但可以包含其他序列,例如靶向向量。要制作數據庫,請在Geneious中創建一個新的空文件夾,然后將要測試的序列導入到該文件夾??中。由于研究人員可能希望測試的基因組種類繁多,基因組序列可能非常大,并且可能會發布新版本的基因組組合,Geneious不包含任何全基因組序列的內置拷貝。可以使用NCBI直接從NCBI下載基因組位于Geneious的Sources面板底部的文件夾(這是Geneious左側的面板)。常用研究的基因組(例如,人類,斑馬魚和大鼠基因組)可以從NCBI中下載,使用Geniouss文件夾中的Geneomes文件夾中的鏈接。基因組也可以從其他來源下載并使用通用文件格式導入Geneious。
在本教程中,已經為您創建了脫靶序列的數據庫。由于本教程確定了釀酒酵母?LYP1基因中的CRISPR位點,我們對釀酒酵母基因組中的脫靶結合感興趣?。釀酒酵母的完整基因組已從NCBI下載并置于酵母基因組文件夾中。要查看這些序列,請打開此文件夾。
要運行Find CRISPR位點工具來識別CRISPR位點并檢查脫靶綁定,請選擇LYP1 CDS文檔(geneious的demo數據,網上提供下載)并轉到克隆→查找CRISPR位點。
在Binding位點旁邊,依次選擇Anywhere。在目標類型“GN(19)”?旁邊的字段以及PAM Site字段中鍵入“NGG”。通過檢查目標活動保留分數位點。
現在通過脫靶分析和分數數據庫來評分分數位點。然后選擇包含我們想測試的序列的文件夾以進行非目標結合:點擊選擇一個文件夾并選擇“酵母基因組”文件夾。對于速度和過濾策略,請選擇慢速 - 對所有位點進行評分。將最大不匹配設置保留為缺省值3,以便與脫離目標不匹配,并允許0為indels。
注意:如果我們沒有選擇Score作為脫離目標數據庫,但選擇在較大序列的選定子區域內查找位點,Geneious將自動測試序列的未選定區域以進行脫靶結合。
這一次,我們希望以偏離目標分數為結果軌跡著色,因此請確保此選項在Color CRISPR sites by設置。對話框現在應該如下圖所示。點擊確定運行搜索。


CRISPR位點查找工具運行后,您將看到以下消息:
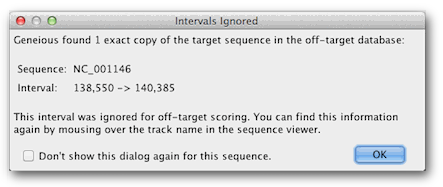

此消息出現是因為我們在LYP1 CDS中搜索CRISPR位點的序列也存在于脫靶數據庫中,因為它是酵母基因組的一部分。由于它們很可能成為本身,因此該區域內的匹配被忽略,因為它們可能是向導位點guide site本身。
與之前的練習一樣,Geneious在LYP1序列中注釋了41個“GN(20)GG”CRISPR位點,并將它們歸入CRISPR Sites。
序列中的每個CRISPR位點已根據其可能結合的脫靶位點數量以及脫靶位點與原序列的相似程度進行評分。“非目標分數”是根據麻省理工學院張峰實驗室開發的方法計算出來的(欲了解更多信息,請點擊這里)。根據每個脫離目標位點與原始CRISPR位點的相似程度以及發生何種不匹配(PAM位點附近的不匹配將比距離PAM位點更遠的不匹配更多地影響綁定)給出評分。脫靶位點的較高分數表明與原始CRISPR位點更高的相似性(并且因此CRISPR / Cas復合物與脫靶標結合的可能性更高)。CRISPR位點的總分是100%減去目標基因組中偏離分數的加權總和。因此,更高的分數表示更好的CRISPR位點,而潛在的異地目標很少或很弱。
序列中的CRISPR注釋現在根據其CRISPR分數進行著色。此配色方案使用紅色到綠色的漸變色,CRISPR分數為20%時注釋為純紅色,85%為純黃色,100%時為純綠色。
單擊保存按鈕將注釋軌道保存在序列中。
打開注解選項卡。要僅顯示注釋表中的CRISPR注釋,請單擊“?類型”并選擇“?CRISPR”。單擊列并勾選脫離目標分數和#脫離目標位點(這些可能已被選中)。這些列現在應該在注釋表中可見。
單擊注釋表中列的名稱將按該列中的值對表中的行進行排序。列名旁邊將出現一個小三角形,指示行是從最小值到最大值進行排序,反之亦然。再次單擊列名將顛倒行排序的方向。按照最低到最高的脫靶評分對注釋表的行進行排序。
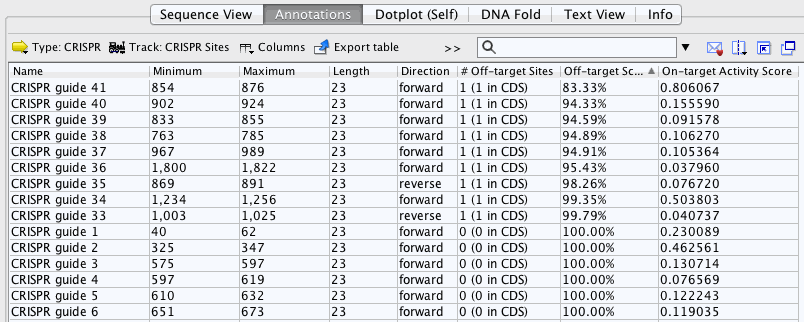

從這張表中你可以看到許多CRISPR位點的偏離目標得分為100%,這意味著它們沒有脫離匹配的匹配符合無插入的條件和3個或更少的與gRNA錯配(但是它們可能有額外的3個以上不匹配的脫靶點)。CRISPR guide 41具有最低的脫靶評分。在表格中選擇此行并返回序列視圖。現在應該在序列視圖中選擇“CRISPR guide 41”注釋。將鼠標懸停在注釋上,出現一個黃色的工具提示。此工具提示包含有關此CRISPR位點的更多信息。
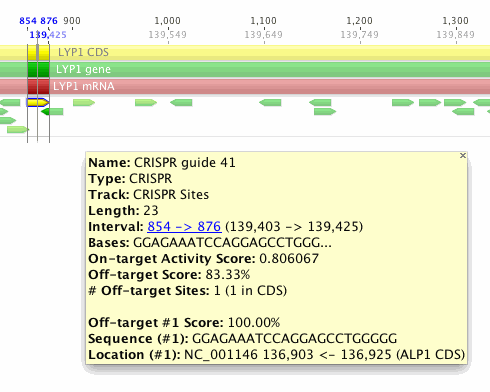

該位點的CRISPR得分為83.33%。釀酒酵母基因組中只有一個此脫序結合位點,但它與CRISPR guide完全匹配,所以脫靶評分為100%。該位點位于136,903位的ALP1 CDS→14號染色體的136,925位。
對于具有多個脫離目標位點的CRISPR guide,只有前五位將列在工具提示中。此信息也可以從“?注釋”選項卡查看,排序和導出。
要從序列中刪除這些注釋,請單擊序列視圖中條目名稱左側的箭頭,然后選擇刪除條目并單擊保存按鈕。
未完,繼續[4]
隨時了解最新的技術快訊
關于這個技術問答社區
Question tools
統計
| 質問: 2018/05/20 14:00 |
| Seen: 7955 times |
| 最終更新: 2018/05/20 14:28 |


